doi: http://dx.doi.org/10.7705/biomedica.v33i2.655
ARTÍCULO ORIGINAL
Contribución de los autores:
Todos los autores participaron por igual en la redacción y producción del manuscrito.
Recibido: 23/02/12; aceptado:06/12/12
Introducción. La linfopoyetina tímica del estroma ( Thymic Stromal Lymphopoietin, TSLP) se ha vinculado como un gen de propensión al desarrollo de enfermedades alérgicas. Se sabe que la población de Cartagena es una mezcla triétnica, en la cual el componente de herencia africana se asoció con el riesgo de asma y altos niveles séricos de IgE total. Este componente provino de esclavos africanos que lograron organizarse en “palenques”, uno de ellos es San Basilio de Palenque, en la Costa Caribe colombiana.
Objetivo. Determinar la distribución de los polimorfismos de nucleótido simple ( Single Nucleotide Polymorphism, SNP) rs1837253, rs17551370 y rs2289276 del gen TSLP en individuos afrodescendientes de San Basilio de Palenque.
Materiales y métodos. Mediante PCR en tiempo real y sondas TaqMan SNP Genotyping ™ se genotipificaron estos SNP en 80 individuos afrodescendientes entre los 5 y 18 años de edad.
Resultados. El alelo de menor frecuencia para el polimorfismo rs1837253 fue el alelo T (41,9 %), para el rs17551370, el alelo A (14,3 %), y para el rs2289276, el alelo T (22,5 %). La distribución de los polimorfismos rs17551370 y rs2289276 se mantuvo en equilibrio genético de Hardy-Weinberg. Las frecuencias alélicas de cada SNP no mostraron diferencias significativas con las reportadas para poblaciones africanas.
Conclusiones. Los tres polimorfismos analizados en el gen TSLP estuvieron presentes en la muestra de población de San Basilio de Palenque y su distribución es similar a la reportada para poblaciones africanas y para poblaciones americanas de ancestro africano.
Palabras clave: frecuencia de los genes, afroamericanos, polimorfismo de nucleótido simple, citocinas, endogamia, Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v33i2.655
Distribution of three polymorphisms of the TSLP gen in African-descendent population from San Basilio de Palenque, Colombia
Introduction: Thymic stromal lymphopoietin (TSLP) has been linked as a susceptibility gene for the development of allergic diseases. It is known that the population of Cartagena is a triethnic mix, in which the component of African ancestry was significantly associated with risk of asthma and high total serum IgE levels. This component comes from African slaves brought into the continent and settled in “palenques”, one of them is San Basilio de Palenque, in the Colombian Caribbean Coast.
Objective: To analyze the distribution of single nucleotide polymorphisms (SNP) rs1837253, rs17551370 and rs2289276 located in TSLP gene, in the African-descendent population of San Basilio de Palenque.
Materials and methods: By real time-PCR and probes TaqMan SNP Genotyping ™ , we genotyped three polymorphisms in 80 individuals of African-descent aged 5 to 18 years of age.
Results: The frequency of the rs1837253 allele T was 41.9%, for the allele A, 14.3% for rs17551370, and 22.5% for the allele T of rs2289276. The rs17551370 and rs2289276 distribution remained in Hardy- Weinberg genetic equilibrium. The allele frequency of each SNP did not show statistically significant differences with those reported for other African and African-descendent populations.
Conclusion: The three polymorphisms in the TSLP were present in the sample population of San Basilio de Palenque and its distribution is similar to that reported for African populations and African ancestry in America.
Key words: gene frequency, African American; polymorphism, single nucleotide; cytokines, inbreeding, Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v33i2.655
Las enfermedades alérgicas son consideradas complejas y multifactoriales, en las cuales intervienen factores genéticos y ambientales. Aún se conoce muy poco sobre las causas iniciales que las desencadenan. Sin embargo, en diversos estudios se demuestra que algunas moléculas de la inmunidad innata son importantes en la desviación hacia una respuesta de linfocitos de tipo Th2 en individuos genéticamente predispuestos. La linfopoyetina tímica del estroma (Thymic Stroma Lymphopoietin, TSLP) es una citocina epitelial que recientemente ha sido involucrada en el inicio de la respuesta inflamatoria de tipo alérgico (1). Esta citocina influye directamente en el desarrollo de las células dendríticas mieloides CD11c+.
Estas células inducen a los linfocitos T vírgenes a diferenciarse hacia un fenotipo Th2, caracterizado por la producción de citocinas inflamatorias, como la IL-4, IL-5, IL-13, y de quimiocinas que inducen la migración de linfocitos T, neutrófilos y eosinófilos al tejido lesionado. De esta manera, la TSLP recrea un ambiente propicio para el desarrollo de procesos inflamatorios de tipo alérgico (1,2). Clínicamente se han observado niveles elevados de dicha citocina en muestras de piel de individuos con dermatitis atópica (3), pulmón de pacientes asmáticos (4), mucosa y pólipos nasales de sujetos con rinitis alérgica (5,6) y córnea de pacientes con queratoconjuntivitis alérgica (7).
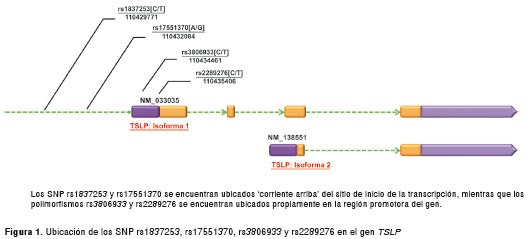
En el humano, el gen que codifica para esta citocina también se denomina TSLP y se ubica en la región cromosómica 5q22.1, se encuentra constituido por cuatro regiones codificadoras y consta de 6.333 bases de longitud. Un splicing alternativo de este gen permite identificar dos isoformas: la isoforma larga, que corresponde a la citocina funcional, y la isoforma corta, que pierde los primeros dos exones 5´ pero contiene un exón 5´ alternativo que difiere en la región 5´ÚTR de la isoforma larga, debido a que emplea un codón de inicio de la transcripción situado dentro del marco de lectura (figura 1).

Recientemente, en diversos estudios se han asociado variantes genéticas presentes en el gen TSLP con la propensión a las enfermedades alérgicas. Harada, et al ., identificaron 23 polimorfismos de nucleótido simple (Single Nucleotide Polymorphism, SNP) en individuos asmáticos. El SNP rs3806933 que se encuentra situado en la región promotora de TSLP , crea un sitio de unión para la proteína activadora 1 (Activator Protein 1, AP-1), factor de transcripción que parece contribuir en la respuesta inmunitaria de tipo Th2, lo que provoca la síntesis de altos niveles de TSLP, como se observó in vitro en células de epitelio bronquial humano en respuesta a estímulos microbianos (8). Hunninghake, et al., reportaron que el alelo T del SNP rs2289276 se asoció con niveles bajos de IgE específica contra cucaracha e IgE total, en niñas asmáticas de una población costarricense con ancestro español y amerindio (9). Gao, et al ., observaron una asociación estadísticamente significativa entre el polimorfismo rs2289276 y la dermatitis atópica en 339 individuos afroamericanos (10). Recientemente, Harada, et al., describieron la asociación de los polimorfismos rs3806933 y rs2289276 con propensión a atopia en niños y asma en adultos japoneses (11).
Por otra parte, He, et al ., reportaron un efecto protector del SNP rs1837253 en el desarrollo de asma, asma atópica e hiperreacción de la vías aéreas, en más de cinco mil individuos canadienses pertenecientes a cuatro diferentes estudios (12). De igual manera, Hunninghake, et al ., observaron que en individuos hispanos, afroamericanos y europeos, el alelo T de rs1837253 se asoció con un menor riesgo de asma en hombres, mientras que el alelo T de rs2289276 se asoció con un menor riesgo en mujeres (13).
En estas mismas poblaciones, Bunyavanich, et al ., demostraron un riesgo reducido de rinitis alérgica en niños asmáticos (14). Por otra parte, Gao, et al., reportaron que el polimorfismo rs17551370 se asoció significativamente con los niveles séricos de IgE total en población afroamericana. Además, demostraron por desequilibrio de ligamiento, que estos SNP anteriormente mencionados se segregan conjuntamente (10).
Según lo anterior, las variantes genéticas presentes en el gen TSLP son de gran interés en cuanto al estudio de las enfermedades alérgicas en diferentes poblaciones. Cabe destacar que la mayoría de las poblaciones a nivel mundial son productos de mezclas recientes, como es el caso de las del continente americano (15,16); dichas mezclas pueden contribuir a la posibilidad de padecer enfermedades complejas, como es el caso de las alergias y fenotipos relacionados como los niveles séricos de IgE total, IgE específica o atopia (17,18).
En Colombia, no existe información sobre la distribución de polimorfismos en el gen TSLP , lo cual es esencial para cualquier estudio de asociación entre variantes genéticas y enfermedades alérgicas. Actualmente, se tiene conocimiento de que la población de Cartagena (Colombia) es una mezcla triétnica entre caucásicos españoles, negros africanos y, en menor proporción, nativos americanos. Previamente, evaluamos la distribución de algunos alelos del complejo HLA y se encontró en un alto porcentaje alelos relacionados con poblaciones caucásicas y africanas (19). Recientemente, se ha podido establecer que el componente de herencia africana en la población de Cartagena se asociaba significativamente con el riesgo de asma y altos niveles séricos de IgE total (20).
Por tales motivos, los estudios genéticos que se hagan en poblaciones fundadoras que contribuyeron en el mestizaje de las actuales poblaciones iberoamericanas, tendrán repercusiones en el conocimiento antropológico y médico. San Basilio de Palenque es una población fundada en la época de la Colonia por esclavos africanos que ingresaron al continente americano por el puerto de Cartagena, durante el comercio esclavista a mediados del siglo XVI, y escaparon del yugo español a finales de este mismo siglo, contribuyendo en su momento en el mestizaje con españoles y amerindios de la época (19).
El objetivo de este estudio consistió en determinar la distribución de los SNP rs1837253, rs17551370 y rs2289276 del gen TSLP , en individuos afrodescendientes de San Basilio de Palenque.
Materiales y métodos
Población de estudio
Se determinó el genotipo de 80 individuos afrodescendientes, entre los 5 y los 18 años de edad, cuyos padres y abuelos eran oriundos del corregimiento de San Basilio de Palenque. Estos sujetos procedían de una cohorte de 200 individuos en quienes previamente se habían evaluado los patrones nutricionales y el estilo de vida (21,22). Debido a la endogamia presente en esta comunidad, solo los 80 sujetos seleccionados para este estudio estaban menos relacionados entre sí, teniendo en cuenta el grado de consanguinidad hasta la tercera generación.
El presente estudio fue aprobado por el Comité de Ética de la Universidad de Cartagena. Los acudientes o padres de todos los participantes firmaron un consentimiento informado, en el que autorizaron la obtención de muestras de sangre, además de aprobar su uso para estudios de investigación.
Extracción de ADN
El ADN se extrajo a partir del sobrenadante ( buffycoat ) de sangre anticoagulada recolectada en tubos de vidrio con EDTA dipotásico y usando el protocolo de salting out y precipitación con perclorato de sodio (23). La concentración de ADN se midió mediante elespectrofotómetro Nanodrop 2000/2000c ™ (Thermo Scientific, Waltham, MA, USA); después, se obtuvieron soluciones de trabajo a una concentración final de 10 ng/µl, las cuales se almacenaron a -20 °C para su uso posterior.
Genotipificación
La genotipificación de los polimorfismos rs1837253, rs17551370 y rs2289276, se hizo mediante reacción en cadena de la polimerasa (PCR) en tiempo real, utilizando los estuches comerciales TaqMan SNP Genotyping Assay ™(Assay ID: C_11910823_20, C_33155477_10 y C_3166723_20, respectivamente) adquiridos de la casa Applied Biosystems, Foster City, CA, USA. Se colocaron 2,4 µl de ADN a una concentración aproximada de 10 ng/µl de cada una de las muestras en pozos individuales de microplacas de 96 pozos, en cada uno de los cuales se agregaron 2,5 µl de Master Mix-2x y 0,125 µl de sondas Taqman Genotyping - 40x específicas para cada SNP. Todas las reacciones de PCR se hicieron por duplicado con un volumen final de 5 µl por pozo, empleando el equipo ABI Prism 7300 de Applied Biosystems, el cual se programó con un paso inicial de 10 minutos a 95 °C, seguido por 40 ciclos, cada uno de 15 segundos a 92 °C y un minuto a 60 °C.
Mediante la aplicación Allelic Discrimination de Applied Biosystems , se determinó la asignación automática de los genotipos resultantes a partir de la calidad de la amplificación de cada muestra con valores de 90 % o más; se repitieron las muestras en las que no se hizo la asignación automática o cuya calidad de amplificación fue inferior al 90 %.
Análisis estadístico
Las frecuencias genotípicas y alélicas se calcularon utilizando el software SPSS ™ ( Statistical Package for the Social Sciences ), versión 17 para Windows. El equilibrio de Hardy-Weinberg se determinó mediante la prueba de asociación no aleatoria refinada con el método de Markov-Chain del software Arlequin, versión 3.5.1.2 (24). Mediante la prueba exacta de diferenciación de poblaciones presente en este mismo software , se pudo comparar la distribución de los alelos evaluados con las frecuencias alélicas de otras poblaciones reportadas en la literatura científica.
Resultados
La edad promedio de los individuos vinculados al estudio fue de 12 años. El 47,5 % (n=38) de los participantes correspondió a niños nacidos en Cartagena de Indias, mientras que el 52,5 % (n=42) restante eran niños originarios del corregimiento de San Basilio de Palenque. Cabe destacar que los padres y los abuelos de todos los participantes eran o son oriundos de esta comunidad afrodescendiente. Por otra parte, la proporción de sujetos de sexo femenino y masculino fue semejante: 48,3 % (n=39) y 51,3 % (n=41), respectivamente.
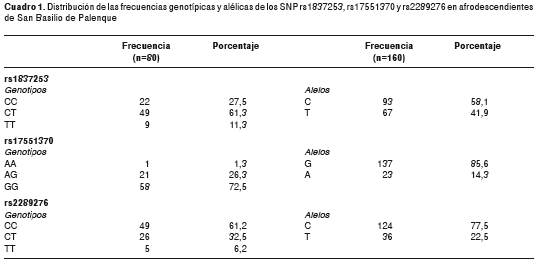
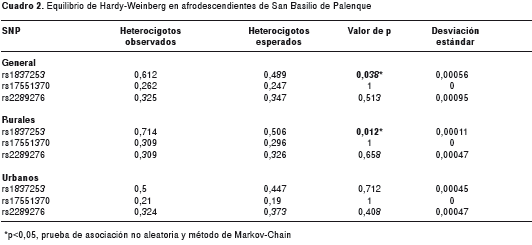
En el cuadro 1 se presenta la distribución genotípica de los SNP rs1837253, rs17551370 y rs2289276 del gen TSLP en los 80 participantes del estudio. Con respecto a la distribución de los alelos, en el polimorfismo rs1837253, el de menor frecuencia fue el alelo T (41,9 %), en el rs17551370, fue el alelo A (14,3 %), y en el rs2289276, fue el alelo T (22,5 %) (cuadro 1). Mediante la prueba de asociación no aleatoria refinada con el método de Markov-Chain, solo los SNP rs17551370 y rs2289276 se encontraron en equilibrio genético de Hardy-Weinberg (p=1,00 y 0,51; respectivamente), a diferencia de la variante rs1837253 (p=0,038) (cuadro 2).
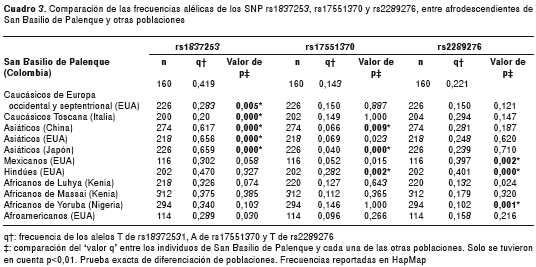
La prueba exacta de diferenciación de población permitió comparar las frecuencias alélicas observadas en individuos afrodescendientes de San Basilio de Palenque y las frecuencias reportadas en poblaciones caucásicas, asiáticas, africanas, hispanas y afroamericanas, disponibles en la base de datos del consorcio internacional HapMap (25).
Como se muestra en el cuadro 3, la frecuencia alélica del polimorfismo rs1837253 se diferenció significativamente (p<0,01) con poblaciones caucásicas de Europa occidental y septentrional, además de poblaciones asiáticas como China y Japón. Algo similar se observó con la frecuencia del SNP rs17551370, cuya diferencia se presentó con poblaciones asiáticas e hindúes. El rs2289276 fue el único que mostró diferencias con una población africana, como es Nigeria, sin dejar de lado poblaciones con descendencia de India y México.
Dada la disposición conjunta de individuos oriundos del ambiente urbano (Cartagena), así como del ambiente rural (San Basilio de Palenque), y teniendo en cuenta que todos los sujetos presentan características genéticas similares, se analizó la distribución de estos tres polimorfismos según el ambiente de procedencia. La distribución genotípica y alélica de los SNP rs1837253, rs17551370 y rs2289276 entre los individuos urbanos y rurales, se muestra en el cuadro 4. Al comparar la distribución genotípica y alélica de estos polimorfismos entre ambos grupos, solo el SNP rs1837253 mostró una diferencia estadísticamente significativa entre los individuos de ambos ambientes (p=0,02 y 0,03, respectivamente) (cuadro 4). Al analizar las dos muestras, se encontró que la distribución de rs17551370 y rs2289276 se mantuvo en equilibrio genético de Hardy-Weinberg en individuos tanto del ambiente urbano como del rural, mientras que el SNP rs1837253 solo estuvo en equilibrio en los afrodescendientes del ambiente urbano (cuadro 2).

Discusión
Este estudio es el primero en Colombia que describe la distribución genotípica y alélica de tres variantes genéticas del gen TSLP en una muestra de individuos afrodescendientes de la Costa Caribe colombiana. La población de San Basilio de Palenque es una comunidad rural poblada por afrodescendientes, ubicada a 55 km de Cartagena que, desde la época de la Colonia, ha mantenido una identidad cultural propia evitando en gran medida el mestizaje con otras etnias. Este fenómeno, sumado al aislamiento, posiblemente ha permitido mantener intacto el componente genético de esta comunidad, convirtiéndola en una población privilegiada para el desarrollo de estudios antropológicos y genéticos que sirven de referencia o guía sobre las poblaciones actuales.
El tamaño de la muestra fue la principal limitación de la presente investigación, debido al alto grado de endogamia que se encuentra en la comunidad de palenque. Sin embargo, gracias a la ayuda de personas conocedoras de las familias y la comunidad en general, se pudieron escoger 80 individuos en tercer grado de consanguinidad, a partir de 200 sujetos seleccionados al azar que habían participado en estudios previos (21,22). Es posible que los resultados reportados en este estudio no puedan extrapolarse a la población general de San Basilio de Palenque, debido a las limitaciones de la muestra; sin embargo, sí servirán de referencia metodológica para el desarrollo de futuros estudios genéticos en esta comunidad.
Entrando en materia, diversos estudios lingüísticos, socioantropológicos y genéticos han señalado que los afrodescendientes de San Basilio de Palenque procedieron principalmente del occidente y la costa sur del continente africano, como las poblaciones de habla bantú, Angola, Bengala y Congo, entre otras (26). A su vez, este componente africano constituyó una importante influencia genética en el inicio de la población de Cartagena de Indias (27). Los resultados aquí observados no demuestran diferencias significativas entre las distribuciones de rs1837253 y rs17551370 en los sujetos de estudio y las comunidades africanas de Yoruba en Nigeria, y Luhya y Massai en Kenia, así como también con individuos afrodescendientes norteamericanos. Sin embargo, la distribución de rs2289276 mostró diferencias con la población de Yoruba, a pesar de ser una comunidad del África occidental. Este hallazgo posiblemente se deba a que las comunidades actuales presentes en Nigeria y otros países del occidente africano, como Camerún, Guinea Ecuatorial y Gabón, son producto de migraciones de poblaciones desde territorios del este y el centro de África (28). Esto ha generado mezclas desde el punto de vista lingüístico y genético, que pueden verse reflejadas como discrepancias en la distribución de marcadores genéticos en las comunidades afrodescendientes del continente americano.
Por otra parte, con los hallazgos observados se podría explicar parte del estado actual de San Basilio de Palenque como sociedad. Según reportes del DANE (Departamento Administrativo Nacional de Estadística, Colombia) del año 2005, la comunidad palenquera se encuentra en una transición demográfica hacia centros urbanos, donde el 63 % (n=4.708) del total de la población habita en ciudades como Cartagena y Barranquilla, mientras que el 37 % (n=2.762) restante aun permanece en el corregimiento de San Basilio de Palenque.
En este estudio se tuvo la posibilidad de evaluar individuos afrodescendientes de esta comunidad que habitan en ambos ambientes. Todo este proceso migratorio podría estar alterando la distribución de marcadores genéticos como el SNP rs1837253, la cual fue diferente entre individuos del ambiente rural y del urbano. Es posible que se estén presentando mezclas aisladas entre palenqueros e individuos de comunidades aledañas a San Basilio de Palenque, especialmente en los afrodescendientes rurales.
Es bien sabido que la prevalencia de las enfermedades alérgicas viene en aumento desde la última década (29). En Colombia estas enfermedades afectan a más del 20 % de la población y en reportes preliminares se sugiere un incremento de sus cifras en los últimos años (Caraballo LR, comunicación personal). Como ya se ha mencionado, el gen TSLP se ha vinculado como un gen de propensión al desarrollo de estas enfermedades y demás alteraciones relacionadas, en poblaciones hispanas, afroamericanas y europeas (13,14). Por tal motivo, todos los estudios genéticos que vinculen polimorfismos de este gen, permitirán entender más a fondo los mecanismos que regulan la expresión y los efectos de esta citocina en el desarrollo de la respuesta inmunitaria hacia un fenotipo Th2 como es el caso de los estudios de asociación y epigenéticos, así como también, inferir la posible influencia de estas variaciones genéticas en poblaciones de mezcla reciente como la de Cartagena de Indias.
A partir de este estudio piloto se concluye que los polimorfismos rs1837253, rs17551370 y rs2289276 del gen TSLP estuvieron presentes en individuos afrodescendientes de San Basilio de Palenque y su distribución es similar a la reportada para poblaciones africanas y de herencia africana en América. Es importante continuar caracterizando genes implicados en enfermedades, ya que se trata de una población que recién empieza a perder su identidad cultural y genética debido a mezclas aisladas con individuos de comunidades aledañas, al crecimiento de la población y la migración a centros urbanos.
Los autores agradecen a la Escuela Benkos Bioho del San Basilio de Palenque, a Regina Miranda y Mayra Salinas pertenecientes a esta comunidad, quienes ayudaron voluntaria e incondicionalmente en el desarrollo de esta investigación. También, a Colciencias por su programa de joven investigador, que favoreció con una beca-pasantía a Luis Fang Mercado.
Los autores del artículo hacen constar que no existe, de manera directa o indirecta, ningún tipo de conflicto de intereses financieros, académicos o personales, que puedan poner en peligro la validez de la investigación.
La financiación de este proyecto fue asumida por la Universidad de Cartagena, según resolución N° 4396 de 2010.
Correspondencia: Javier Marrugo, Instituto de Investigaciones Inmunológicas, Universidad de Cartagena, Campus de Zaragocilla, edificio de la biblioteca, primer piso. Cartagena, Colombia Telefax: (575) 669 8491 jmarrugoc@unicartagena.edu.co
Referencias1. Ito T, Liu YJ, Arima K. Cellular and molecular mechanisms of TSLP function in human allergic disorders –TSLP programs the “Th2 code” in dendritic cells. Allergol Int. 2012;61:35-43. http://dx.doi.org/10.2332/allergolint.11-RAI-0376
2. Comeau MR, Ziegler SF. The influence of TSLP on the allergic response. Mucosal Immunol. 2009;3:138-47. http://dx.doi.org/10.1038/mi.2009.134.
3. Soumelis V, Reche PA, Kanzler H, Yuan W, Edward G, Homey B, et al. Human epithelial cells trigger dendritic cell mediated allergic inflammation by producing TSLP. Nat Immunol. 2002;3:673-80. http://dx.doi.org/10.1038/ni805.
4. Ying S, O´Connor B, Ratoff J, Meng Q, Mallett K, Cousins D, et al. Thymic stromal lymphopoietin expression is increased in asthmatic airways and correlates with expression of Th2-attracting chemokines and disease severity. J Immunol. 2005;174:8183-90.
5. Mou Z, Xia J, Tan Y, Wang X, Zhang Y, Zhou B, et al. Overexpression of thymic stromal lymphopoietin in allergic rhinitis. Acta Otolaryngol. 2009;129:297-301. http://dx.doi.org/10.1080/00016480802225884.
6. Kimura S, Pawankar R, Mori S, Nonaka M, Masuno S, Yagi T, et al . Increased expression and role of thymic stromal lymphopoietin in nasal polyposis. Allergy Asthma Immunol Res. 2011;3:186-93. http://dx.doi.org/10.4168/aair.2011.3.3.186.
7. Matsuda A, Ebihara N, Yokoi N, Kawasaki S, Tanioka H, Inatomi T, et al. Functional role of thymic stromal lymphopoietin in chronic allergic keratoconjunctivitis. Invest Ophthalmol Vis Sci. 2009;51:151-5. http://dx.doi.org/10.1167/iovs.09-4183.
8. Harada M, Hirota T, Jodo AI, Doi S, Kameda M, Fujita K, et al . Functional analysis of the thymic stromal lymphopoietin variants in human bronchial epithelial cells. Am J Respir Cell Mol Biol. 2009;40:368-74. http://dx.doi.org/10.1165/ rcmb.2008-0041OC.
9. Hunninghake GM, Lasky-Su J, Soto-Quiros ME, Avila L, Liang C, Lake SL, et al . Sex-stratified linkage analysis identifies a female-specific locus for IgE to cockroach in Costa Ricans. Am J Respir Crit Care Med. 2008;177:830-6. http://dx.doi.org/10.1164/rccm.200711-1697OC
10. Gao PS, Rafaels NM, Mu D, Hand T, Murray T, Boguniewicz M, et al . Genetic variants in thymic stromal lymphopoietin are associated with atopic dermatitis and eczema herpeticum. J Allergy Clin Immunol. 2010;125:1403-7. http://dx.doi.org/10.1016/j.jaci.2010.03.016
11. Harada M, Hirota T, Jodo AI, Hitomi Y, Sakashita M, Tsunoda T, et al . Thymic stromal lymphopoietin gene promoter polymorphisms are associated with susceptibility to bronchial asthma. Am J Respir Cell Mol Biol. 2011;44:787- 93. http://dx.doi.org/10.1165/rcmb.2009-0418OC
12. He JQ, Hallstrand TS, Knight D, Chan-Yeung M, Sandford A, Tripp B, et al. A thymic stromal lymphopoietin gene variant is associated with asthma and airway hyperresponsiveness. J Allergy Clin Immunol. 2009;124:222-9. http://dx.doi.org/10.1016/j.jaci.2009.04.018
13. Hunninghake GM, Soto-Quiros ME, Ávila L, Kim HP, Lasky-Su J, Rafaels N, et al. TSLP polymorphisms are associated with asthma in a sex-specific fashion. Allergy. 2010;65:1566-75. http://dx.doi.org/10.1111/j.1398- 9995.2010.02415.x
14. Bunyavanich S, Melen E, Wilk JB, Granada M, Soto-Quirós ME, Ávila L, et al. Thymic stromal lymphopoietin (TSLP) is associated with allergic rhinitis in children with asthma. Clin Mol Allergy. 2011;9:1. http://dx.doi.org/10.1186/1476-7961-9-1.
15. Bortolini MC, Salzano FM, Thomas MG, Stuart S, Nasanen SP, Bau CH, et al. Y-chromosome evidence for differing ancient demographic histories in the Americas. Am J Hum Genet. 2003;73:524-39.
16. Acosta MA, Blanco-Verea A, Lareu MV, Brion M, Carracedo A. The genetic male component of two South-Western Colombian populations. Forensic Sci Int Genet. 2009;3:59- 61. http://dx.doi.org/10.1016/j.fsigen.2008.06.006
17. Sharma S, Poon A, Himes BE, Lasky-Su J, Sordillo JE, Belanger K, et al. Association of variants in innate immune genes with asthma and eczema. Pediatr Allergy Immunol. 2012;23:315-23. http://dx.doi.org/10.1111/j.1399-3038.2011.01243.x
18. Vercelli D. Discovering susceptibility genes for asthma and allergy. Nat Rev Immunol. 2008;8:169-82. http://dx.doi.org/10.1038/nri2257
19. Caraballo LR, Marrugo J, Erlich H, Pastorizo M. HLA alleles in the population of Cartagena (Colombia). Tissue Antigens. 1992;39:128-33.
20. Vergara C, Caraballo L, Mercado D, Jiménez S, Rojas W, Rafaels N, et al . African ancestry is associated with risk of asthma and high total serum IgE in a population from the Caribbean Coast of Colombia. Hum Genet. 2009;125:565-79. http://dx.doi.org/10.1007/s00439-009-0649-2
21. Marrugo JA, Urrego JR, Fang LC, Munoz CA, Hernández LC, López L. Influence of rural and urban dietary and lifestyle patterns in atopic diseases in a Colombian African-descent population. J Allergy Clin Immunol. 2012;129:AB130.
22. Fang L, Urrego J, Muñoz C, Hernández L, Lopez-Kleine L, Marrugo J. Little changes in a monotone dietary pattern may influence in atopy in African-descendent Colombian children. Clinical Nutrition Supplements. 2012;7:171.
23. Johns MB, Jr., Paulus-Thomas JE. Purification of human genomic DNA from whole blood using sodium perchlorate in place of phenol. Anal Biochem. 1989;180:276-8. http:// dx.doi.org/10.1016/0003-2697(89)90430-2
24. Excoffier L, Lischer HE. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows. Mol Ecol Resour. 2010;10:564-7. http://dx.doi.org/10.1111/j.1755-0998.2010.02847.x
25. International HapMap Consortium. The International HapMap Project. Nature. 2003;426:789-96.
26. Hiernaux J. Bantu expansion: The evidence from physical anthropology confronted with linguistic and archaeological evidence. J Afr Hist. 1968:505-15.
27. Jiménez S, Martínez B, Hernández M, Múnera A, Caraballo L. Análisis inmunogenético y antropológico de la población del Palenque de San Basilio (Colombia). Polimorfismo génico (HLA) en poblaciones hispanoamericanas. Real Academia de Ciencias Exactas, Físicas y Naturales, Madrid, España. 1996:245-69.
28. Montano V, Ferri G, Marcari V, Batini C, Anyaele O, Destro-Bisol G, et al . The Bantu expansion revisited: A new analysis of Y chromosome variation in Central Western Africa. Mol Ecol. 2011;20:2693-708. http://dx.doi.org/10.1111/j.1365-294X.2011.05130.x
29. Ebert CS Jr, Pillsbury HC 3rd. Epidemiology of allergy. Otolaryngol Clin North Am. 2011;44:537-48. http://dx.doi.org/10.1016/j.otc.2011.03.001